机器学习异常值识别和处理方法(附代码)
type
Post
date
Nov 2, 2023
summary
异常值可能是在数据采集、数据记录、数据提取等步骤中产生的,异常值的出现会对模型学习进行干扰,所以需要进行特殊处理。而在实际的生产环境中的异常值更是五花八门。
category
学习笔记
tags
异常值
特征工程
数据预处理
箱型图
异常检测
机器学习
四分位
password
URL
Property
Jun 20, 2025 01:47 AM
异常值是远离其他数据点的数据点。换句话说,它们是一个数据集中不同寻常的值。异常值对于许多统计分析是有问题的,因为它可能让我们错过显著发现或扭曲实际结果。
一、单变量异常检测
1.1、四分位距法(箱型图)
四分位距(Interquartile Range, IQR)是一种常用的统计方法来检测异常值。它基于数据的四分位数,即将数据分为四等份的值。其中,第一四分位数(Q1)和第三四分位数(Q3)之间的距离称为四分位距。异常值通常定义为小于Q1 - 1.5 * IQR或大于Q3 + 1.5 * IQR的值。
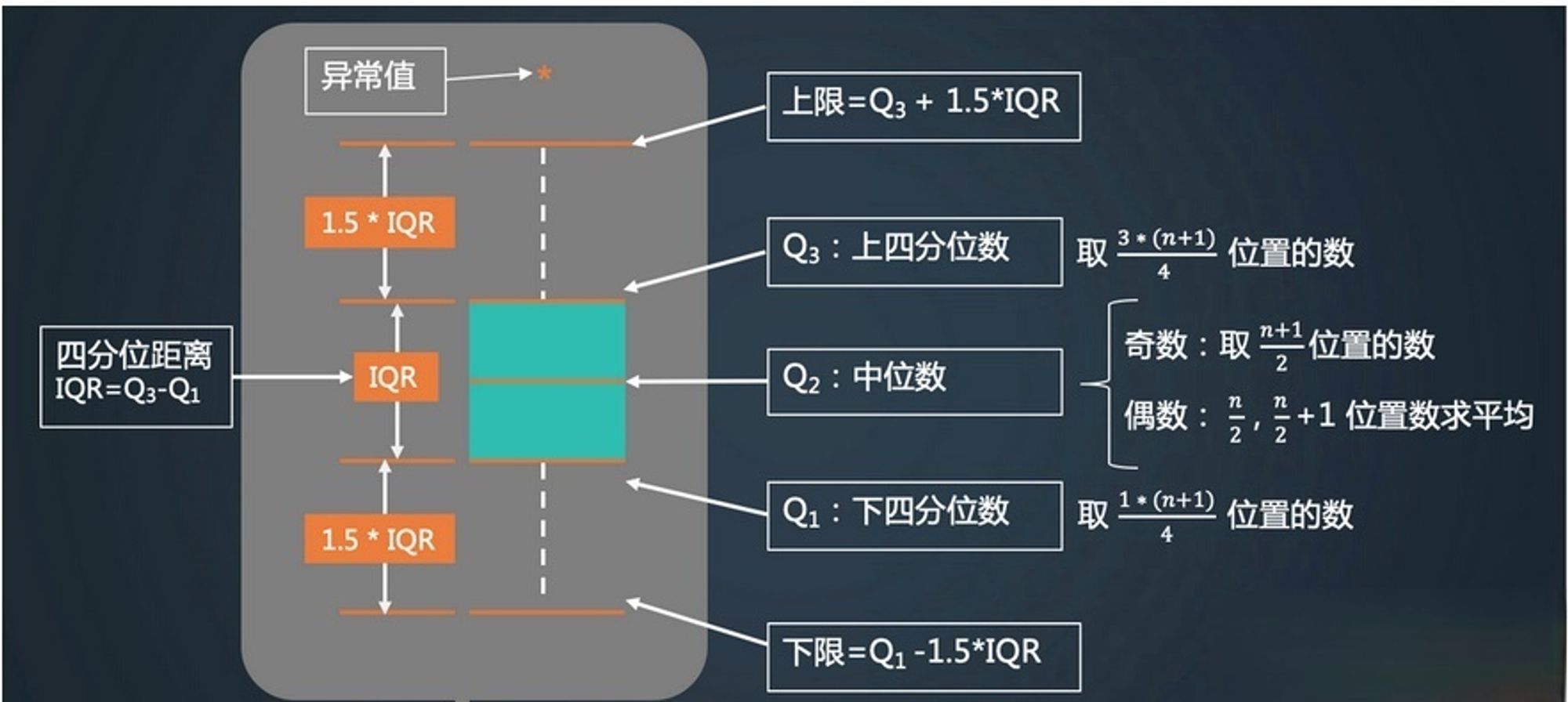
箱形图的上限和下限并不是数据集的最大值和最小值,而是经过一定的计算得到的,所以能检测到离群比较离谱的异常值
在 pandas 中,可以用 quantile 方法来计算指定的分位数,并结合箱型图的特性:1.5 倍四分位距离的特性来过滤异常值(可以适当调整倍数或分位数)
numpy实现,识别单组数据的异常值
import numpy as np def detect_outliers(data): """ 使用四分位距法检测异常值 参数: data: 输入的数据数组 返回: outliers: 检测到的异常值列表 """ # 计算四分位数 Q1 = np.percentile(data, 25) Q3 = np.percentile(data, 75) IQR = Q3 - Q1 # 计算异常值的阈值 lower_bound = Q1 - 1.5 * IQR upper_bound = Q3 + 1.5 * IQR # 检测异常值 outliers = [x for x in data if x < lower_bound or x > upper_bound] return outliers # 示例使用 data = [-10,5, 7, 7, 8, 8, 9, 10, 10, 11, 13, 14, 15, 16, 17, 18, 19, 20, 22, 23, 29, 30, 34, 100] outliers = detect_outliers(data) print("异常值:", outliers) # 绘制箱型图 plt.boxplot(data, vert=False, patch_artist=True) # vert=False使箱型图水平显示 plt.title("boxplot") plt.xlabel("value") plt.show()
pandas实现,识别每一列的异常值
import pandas as pd def detect_outliers_df(df): """ 使用四分位距法检测DataFrame中每一列的异常值 参数: df: 输入的DataFrame 返回: outliers_dict: 一个字典,包含每一列的异常值及其对应的索引 """ outliers_dict = {} cleaned_df = df.copy() for column in df.columns: Q1 = df[column].quantile(0.25) Q3 = df[column].quantile(0.75) IQR = Q3 - Q1 lower_bound = Q1 - 1.5 * IQR upper_bound = Q3 + 1.5 * IQR # 检测异常值 outliers = df[(df[column] < lower_bound) | (df[column] > upper_bound)] # 如果存在异常值,则添加到字典中 if not outliers.empty: outliers_dict[column] = outliers[[column]] cleaned_df = cleaned_df[(cleaned_df[column] >= lower_bound) & (cleaned_df[column] <= upper_bound)] return cleaned_df, outliers_dict # 构造一个有多列的DataFrame数据,每一列中都有一些异常值 data = { 'A': [1, 2, 3, 4, 5, 100], 'B': [10, 20, 30, 40, 50, -100], 'C': [2, 4, 6, 8, 10, 200] } df = pd.DataFrame(data) # 使用函数检测异常值 outliers_df, outliers_dict = detect_outliers_df(df) for column, outliers in outliers_dict.items(): print(f"列 {column} 的异常值及其索引:\n{outliers}\n") print('剔除异常值后的数据:\n',outliers_df)
1.2、标准差法(3σ 原则)
通常一组数据的标准差用 σ 表示,算术平均用 μ 表示。

对于服从正态分布的数据来说,约68.2%的数据在均值的一倍标准差之内。约有95.4%和99.7%的数据点在均值的两倍和三倍标准差以内。所以使用这个方法是在假设数据服从标准正态分布的情况下。
3σ 原则是通过设置一个上限:μ + 3σ;一个下限:μ - 3σ;将分布在这个上下限范围之外的数据视为异常数据。
numpy实现,识别单组数据的异常值
import numpy as np def detect_outliers_std(data): """ 使用3倍标准差法检测异常值 参数: data: 输入的数据数组 返回: outliers: 检测到的异常值列表 """ mean = np.mean(data) std = np.std(data) # 计算异常值的阈值 lower_bound = mean - 3 * std upper_bound = mean + 3 * std # 检测异常值 outliers = [x for x in data if x < lower_bound or x > upper_bound] return outliers # 构造一组数据,其中包含一些异常值 data = [10, 12, 12, 13, 12, 11, 14, 13, 15, 102, 12, 14, 13, 12, 110, 12, 13, 14, 15, 11, 12, 13] outliers = detect_outliers_std(data) print("异常值:", outliers)
pandas实现,识别每一列的异常值
import pandas as pd import numpy as np def detect_and_remove_outliers_std_df(df): """ 使用3倍标准差法检测并移除DataFrame中每一列的异常值 参数: df: 输入的DataFrame 返回: outliers_info: 一个字典,包含每一列的异常值及其对应的索引 cleaned_df: 删除异常值后的DataFrame """ outliers_info = {} cleaned_df = df.copy() for column in df.columns: mean = df[column].mean() std = df[column].std() # 计算异常值的阈值 lower_bound = mean - 3 * std upper_bound = mean + 3 * std # 检测异常值 outliers = df[(df[column] < lower_bound) | (df[column] > upper_bound)] # 如果存在异常值,则添加到字典中 if not outliers.empty: outliers_info[column] = outliers[[column]] # 从cleaned_df中移除这一列的异常值 cleaned_df = cleaned_df[(cleaned_df[column] >= lower_bound) & (cleaned_df[column] <= upper_bound)] return outliers_info, cleaned_df # 创建正态分布数据 np.random.seed(0) data = { 'A': np.random.normal(loc=0, scale=1, size=1000), 'B': np.random.normal(loc=5, scale=2, size=1000), 'C': np.random.normal(loc=10, scale=3, size=1000) } df = pd.DataFrame(data) # 插入异常值 df.loc[100, 'A'] = 10 df.loc[200, 'B'] = -10 df.loc[300, 'C'] = 35 # 使用函数检测并移除异常值 outliers_info, cleaned_df = detect_and_remove_outliers_std_df(df) # 打印异常值信息 for column, outliers in outliers_info.items(): print(f"列 {column} 的异常值及其索引:\n{outliers}\n") # 打印清理后的DataFrame print("清理后前的DataFrame:\n", df.shape) print("清理后的DataFrame:\n", cleaned_df.shape)

1.3、z-score 法
Z分数是一种数学变换,它根据每个观测值与平均值的距离对其进行分类。该方法计算数组中每个值的z-score值,然后再根据设定的阈值来筛选样本,其计算公式为:
z-score 法其实与标准差法的底层逻辑一致,当设定的阈值为 [-3, 3] 时,其实就是 3σ 原则
二、多变量异常检测
2.1、异常检测算法
2.1.1、孤立森林
孤立森林(Isolation Forest)假设异常点是稀有的且和正常数据的分布是偏离的,他的的基本原理是随机选择一个特征和该特征的一个随机切分值来递归地分割数据,异常点通常是稀有的和不同于大部分数据的,因此它们更容易被孤立出来,所以他们被孤立出来的路径会比较短,路径较短的点更可能是异常值。模型输出标记为 -1 的判定为异常值
经验提示:
在对数据进行异常值识别的时候,应该分别对训练数据和预测数据进行训练预测(也就是都用 fit_predict 方法)而不是用训练数据训练一个模型后对新数据数据直接用 predict 进行预测。因为新数据和训练数据的空间分布是不一样的,所以这样识别出来的异常值是不对的。
之前用训练数据训练了一个孤立森林模型,然后在预测的时候直接加载模型来预测,结果预测出来之后异常值和正常值的比例反过来了,标签为 1 的正常值数量看起来倒像是异常值(因为等着部署模型上线当时真的汗流浃背了~)。当时我以为是 fit_predict 方法和 predict 方法返回的标签相反(后来细想官方应该不会干这么 low 的事),结果去查了官网原文,发现是我的结果问题。排查了好久才反应过来是这个问题。
import numpy as np import matplotlib.pyplot as plt from sklearn.ensemble import IsolationForest # 构造一些二维数据,包括正常点和异常点 np.random.seed(42) X_normal = np.random.randn(100, 2) * 2 X_outliers = np.random.uniform(low=-10, high=10, size=(20, 2)) X = np.vstack([X_normal, X_outliers]) # 创建孤立森林模型 clf = IsolationForest(n_estimators=100, contamination=0.15, random_state=42) # 拟合模型 clf.fit(X) # 预测数据点的异常状态 y_pred = clf.predict(X) # 可视化结果 plt.figure(figsize=(10, 6)) plt.scatter(X[y_pred == 1][:, 0], X[y_pred == 1][:, 1], c='green', label='Normal points') plt.scatter(X[y_pred == -1][:, 0], X[y_pred == -1][:, 1], c='red', label='Outliers') plt.title("Isolation Forest Anomaly Detection") plt.xlabel("Feature 1") plt.ylabel("Feature 2") plt.legend() plt.show()
参数详解:
# n_estimators:基分类器的数量;默认100 # max_samples:用来训练基分类器的样本个数或比例; # 值有:auto,int,float; # 如果值是大于0的整数,则抽取整数个样本 # 如果值是浮点数(0到1之间的浮点数),则抽取data.shape[0] * float 个样本 # 如果值是auto,则抽取min(256, n_samples)个样本。n_samples代表所有样本数量 # contamination:数据污染程度,也就是异常值所占的比例。 # 如果值为auto,则默认这个比例和作者论文的数据污染比例一致,默认0.1 # 如果是一个float,则这个float的值应该在(0,0.5)之间 # max_features:用来训练基分类器的特征个数或比例 # 如果值是大于0的整数,则抽取整数个特征 # 如果值是浮点数(0到1之间的浮点数),则抽取data.shape[1] * float 个特征
2.1.2、局部异常因子 LOF
核心思想是通过比较给定数据点与其邻居的局部密度偏差来识别异常点。局部密度是通过计算一个点与其最近邻之间的距离来估计的。如果一个点的局部密度显著低于其邻居,那么这个点就被认为是一个局部离群点。模型输出标记为 -1 的判定为异常值
import numpy as np import matplotlib.pyplot as plt from sklearn.neighbors import LocalOutlierFactor # 构造数据:一个密集的簇和一些离群点 np.random.seed(42) # 确保结果可重复 X_inliers = 0.3 * np.random.randn(100, 2) X_outliers = np.random.uniform(low=-4, high=4, size=(20, 2)) X = np.r_[X_inliers + 2, X_outliers] # 使用LOF算法 clf = LocalOutlierFactor(n_neighbors=20, contamination=0.1) y_pred = clf.fit_predict(X) # 可视化结果 plt.title("Local Outlier Factor (LOF)") # 正常点 plt.scatter(X[y_pred == 1][:, 0], X[y_pred == 1][:, 1], color='blue', s=10, label='Inliers') # 异常点 plt.scatter(X[y_pred == -1][:, 0], X[y_pred == -1][:, 1], color='red', s=10, label='Outliers') plt.axis('tight') plt.xlim((-5, 5)) plt.ylim((-5, 5)) plt.xlabel("Feature 1") plt.ylabel("Feature 2") plt.legend() plt.show()
2.1.3、One class SVM
One-Class SVM是一种无监督学习算法。尽管它属于支持向量机(SVM)家族,但与传统的SVM不同,One-Class SVM不是为了解决分类或回归问题,而是专门设计用于异常检测。One-Class SVM的目标是基于这些正常数据学习一个决策边界,以便能够识别出新的、未见过的数据点是否属于异常。因此,它不需要异常数据的标签,适用于只有单一类别数据可用的情况,这是典型的无监督学习场景。
One-Class SVM的工作原理基于将所有的数据点映射到一个高维空间,并在这个空间中寻找最大的间隔来区分正常点和异常点。
使用正常数据来训练,然后将带有异常样本的数据用来预测。
优点
- 有效处理高维数据:One-Class SVM通过使用核技巧,能够有效处理高维数据。
- 无需异常样本:作为一种无监督学习算法,One-Class SVM不需要异常样本就可以进行训练。
- 灵活性:通过调整参数,可以控制模型对异常的敏感度。
缺点
- 参数选择敏感:算法的性能在很大程度上依赖于参数的选择,如核函数的选择和正则化参数。
- 计算成本高:特别是在处理大规模数据集时,计算成本可能会很高。
- 对数据分布敏感:如果正常数据的分布不是紧凑的,One-Class SVM的性能可能会下降。
import numpy as np import matplotlib.pyplot as plt from sklearn.svm import OneClassSVM # 构造数据集 np.random.seed(42) X_normal = 0.3 * np.random.randn(100, 2) + 2 # 正常样本集中在(2,2)附近 X_outliers = np.random.uniform(low=-4, high=4, size=(20, 2)) # 异常样本分布更广 X = np.r_[X_normal, X_outliers] # 训练One-Class SVM模型 # 通过调整'nu'和'gamma'参数来优化模型 clf = OneClassSVM(nu=0.05, kernel="rbf", gamma=0.5) clf.fit(X_normal) # 预测 y_pred = clf.predict(X) y_pred_normal = y_pred[:100] y_pred_outliers = y_pred[100:] # 绘制正常样本和异常样本 plt.figure(figsize=(10, 6)) plt.title("Optimized One-Class SVM") # 正常样本 plt.scatter(X_normal[:, 0], X_normal[:, 1], c='blue', s=50, edgecolors='k', label='Normal Samples') # 异常样本 # plt.scatter(X_outliers[:, 0], X_outliers[:, 1], c='red', s=50, edgecolors='k', label='Outliers') # 用于预测的样本 plt.scatter(X[y_pred == -1][:, 0], X[y_pred == -1][:, 1], s=100, facecolors='none', edgecolors='r', label='Predicted Outliers') plt.xlabel("Feature 1") plt.ylabel("Feature 2") plt.legend() plt.show()
2.2、聚类算法
2.2.1、K-means
使用 K-means 聚类之后,基于数据点到其所属簇中心的距离来判断是否为异常点。数据点到簇中心的距离远大于大多数点的距离时,这个点可能是异常点。
import numpy as np from sklearn.cluster import KMeans import matplotlib.pyplot as plt # 生成模拟数据 np.random.seed(42) X = np.random.rand(100, 2) # 使用K-Means算法进行聚类,这里K设置为3 kmeans = KMeans(n_clusters=3, random_state=42) kmeans.fit(X) labels = kmeans.labels_ centers = kmeans.cluster_centers_ # 计算每个点到其簇中心的距离 distances = np.linalg.norm(X - centers[labels], axis=1) # 计算距离的平均值和标准差 mean_distance = np.mean(distances) std_distance = np.std(distances) # 设置阈值为平均距离加上两倍的标准差 threshold = mean_distance + 2 * std_distance # 识别异常点 outliers = distances > threshold # 绘制结果 plt.scatter(X[:, 0], X[:, 1], c='blue', marker='o', s=50, label='Normal') plt.scatter(X[outliers, 0], X[outliers, 1], c='red', marker='o', s=50, label='Outliers') plt.scatter(centers[:, 0], centers[:, 1], c='green', marker='x', s=100, label='Centers') plt.title('K-Means Anomaly Detection') plt.xlabel('Feature 1') plt.ylabel('Feature 2') plt.legend() plt.show() # 打印异常点 print("异常点的索引:", np.where(outliers)[0]) print("异常点的坐标:", X[outliers])
2.2.2、DBSCAN
使用聚类算法进行分簇,如果某一个簇里的样本数很少,而且簇质心和其他所有的簇都很远,那么这个簇里面的样本极有可能是异常特征样本了。
DBSCAN 可以识别出高密度区域。算法的结果是一个标签数组,其中
-1表示异常点或噪声。它的优势是不用指定簇的数量。聚类结果说明:
- -1:表示该样本为噪声点或孤立点。它们位于聚类的边缘或空白区域,没有足够的邻居点来形成一个稠密的聚类。
- 0:表示该样本为核心点。核心点是指其周围有足够多的邻居点(即其邻居点数大于等于一个阈值minPts),可以形成一个稠密的聚类。
- 其他正整数:表示该样本属于第几个聚类。DBSCAN将样本划分为多个稠密的聚类,每个聚类都有一个唯一的标签编号。
from sklearn.cluster import DBSCAN import numpy as np import matplotlib.pyplot as plt # 生成两个主要的数据簇 cluster_1 = np.random.normal(loc=[5, 5], scale=1.0, size=(100, 2)) cluster_2 = np.random.normal(loc=[15, 15], scale=1.0, size=(100, 2)) # 生成一些随机的异常点 anomalies = np.random.uniform(low=0, high=20, size=(20, 2)) # 合并数据集 X = np.concatenate([cluster_1, cluster_2, anomalies]) # 应用DBSCAN算法 db = DBSCAN(eps=2, min_samples=5).fit(X) labels = db.labels_ # 打印结果 print("Labels assigned by DBSCAN: ", labels) # 绘制结果 plt.figure(figsize=(10, 6)) plt.scatter(X[:,0], X[:,1], c=labels, cmap='Paired', label='Data points') plt.title('DBSCAN for Anomaly Detection with More Data') plt.xlabel('Feature 1') plt.ylabel('Feature 2') # 标记异常点 for i, txt in enumerate(labels): if txt == -1: # 噪声点 plt.annotate('Anomaly', (X[i,0], X[i,1])) plt.legend() plt.show()
2.2.3、OPTICS
是一种基于密度的聚类算法,是对DBSCAN的优化。不需要预先设定全局的密度阈值(即半径ε),可以识别不同密度的簇。
-1表示异常点或噪声。from sklearn.cluster import OPTICS import numpy as np import matplotlib.pyplot as plt # 设置随机种子以保证结果的可重复性 np.random.seed(0) # 构造三个不同密度的数据簇 cluster_1 = np.random.randn(50, 2) * 0.2 + [1, 1] cluster_2 = np.random.randn(30, 2) * 0.1 + [2.5, 2.5] cluster_3 = np.random.randn(100, 2) * 0.3 + [4, 4] # 构造一些孤立的异常点 outliers = np.random.uniform(low=[0,0], high=[5,5], size=(10, 2)) # 合并数据 X = np.vstack([cluster_1, cluster_2, cluster_3, outliers]) # 使用OPTICS算法,调整参数 optics = OPTICS(min_samples=10, xi=0.05, min_cluster_size=0.1) optics.fit(X) # 可视化结果 plt.figure(figsize=(10, 7)) # 绘制数据点 colors = ['b', 'g', 'r', 'c', 'm', 'y', 'k'] labels = optics.labels_ unique_labels = np.unique(labels) for klass, color in zip(unique_labels, colors): if klass == -1: # -1表示异常点 color = 'k' # 将异常点标记为黑色 Xk = X[labels == klass] plt.plot(Xk[:, 0], Xk[:, 1], color+'o', alpha=0.5, markeredgecolor='k', markersize=5) plt.title('OPTICS Clustering with Adjusted Parameters') plt.xlabel('Feature 1') plt.ylabel('Feature 2') plt.show()
2.3、降维算法
2.3.1、PCA异常检测
PCA 用作异常检测的时候有两种方式:
- 将数据映射到低维特征空间,然后在特征空间不同维度上查看每个数据点跟其它数据的偏差;
- 将数据映射到低维特征空间,然后由低维特征空间重新映射回原空间,尝试用低维特征重构原始数据,看重构误差的大小。
import numpy as np import matplotlib.pyplot as plt from sklearn.decomposition import PCA from sklearn.preprocessing import StandardScaler # 生成二维数据 np.random.seed(0) X_inliers = np.random.randn(100, 2) X_outliers = np.random.uniform(low=-4, high=4, size=(20, 2)) X = np.vstack((X_inliers, X_outliers)) # 标准化数据 X_scaled = StandardScaler().fit_transform(X) # 应用PCA pca = PCA(n_components=2) X_pca = pca.fit_transform(X_scaled) X_inverse = pca.inverse_transform(X_pca) # 计算还原误差 reconstruction_error = np.sum(np.square(X_scaled - X_inverse), axis=1) # 设定一个阈值,高于阈值的点被认为是异常 threshold = np.percentile(reconstruction_error, 95) outlier_index = np.where(reconstruction_error > threshold)[0] # 可视化 plt.scatter(X[:, 0], X[:, 1], color='b', label='Total points') plt.scatter(X[outlier_index, 0], X[outlier_index, 1], color='r', label='Detected outliers') plt.xlabel('Feature 1') plt.ylabel('Feature 2') plt.legend() plt.show()
2.4、深度学习方法
2.4.1、自编码器Autoencoder
使用了一个神经网络来将高维输入编码成低维表示,然后再将它重建还原;算法假设异常点和正常点服从不同的分布,根据正常数据训练出来的Autoencoder,能够将正常样本重建还原,但是却无法将异于正常分布的数据点较好地还原,导致其基于重构误差较大。当重构误差大于某个阈值时,将其标记为异常值。
- 自动编码器相对于 PCA 的优势是:使用非线性激活函数时克服了PCA线性的限制。
import torch import torch.nn as nn import numpy as np import matplotlib.pyplot as plt # 定义自编码器模型 class Autoencoder(nn.Module): def __init__(self): super(Autoencoder, self).__init__() self.encoder = nn.Sequential( nn.Linear(2, 1), nn.ReLU(True) ) self.decoder = nn.Sequential( nn.Linear(1, 2), nn.ReLU(True) ) def forward(self, x): x = self.encoder(x) x = self.decoder(x) return x # 生成数据 np.random.seed(0) x_inliers = 0.3 * np.random.randn(100, 2) + 2 # 正常数据 x_outliers = np.random.uniform(low=-4, high=4, size=(20, 2)) # 异常数据 X = np.concatenate([x_inliers, x_outliers]) X_train = x_inliers # 仅使用正常数据进行训练 # 转换为PyTorch张量 X_tensor = torch.Tensor(X) X_train_tensor = torch.Tensor(X_train) # 实例化模型并定义损失函数和优化器 model = Autoencoder() criterion = nn.MSELoss() optimizer = torch.optim.Adam(model.parameters(), lr=0.01) # 训练模型 num_epochs = 100 for epoch in range(num_epochs): # 前向传播 output = model(X_train_tensor) loss = criterion(output, X_train_tensor) # 反向传播和优化 optimizer.zero_grad() loss.backward() optimizer.step() if (epoch+1) % 10 == 0: print(f'Epoch [{epoch+1}/{num_epochs}], Loss: {loss.item():.4f}') # 使用训练好的自编码器对整个数据集进行重构,并计算重构误差 reconstructed = model(X_tensor).detach().numpy() reconstruction_error = np.sum((X - reconstructed)**2, axis=1) # 定义异常检测的阈值 threshold = np.percentile(reconstruction_error, 84) # 假设异常数据占5% # 根据重构误差判断每个点是否异常 is_outlier = reconstruction_error > threshold # 可视化 plt.scatter(X[is_outlier, 0], X[is_outlier, 1], color='red', label='Outliers') plt.scatter(X[~is_outlier, 0], X[~is_outlier, 1], color='blue', label='Inliers') plt.legend() plt.title('Anomaly Detection with Autoencoder') plt.show()
2.4.2、LSTM时序数据异常检测
LSTM可用于时间序列数据的异常检测:利用历史序列数据训练模型,检测与预测值差异较大的异常点。
import numpy as np import matplotlib.pyplot as plt import torch import torch.nn as nn # 生成正弦波数据作为正常数据 t = np.arange(0, 1000) normal_data = np.sin(0.02*t) + np.random.uniform(-0.05, 0.05, 1000) # 添加一些噪声 # 人为设置一段数据为异常,但在训练时不使用这部分数据 anomaly_data = normal_data.copy() anomaly_data[300:400] = 3 plt.plot(t, normal_data, label='normal_data') plt.plot(t, anomaly_data, label='anomaly_data') plt.title("normal_data and anomaly_data") plt.legend() plt.show() # 数据预处理,创建序列 def create_sequences(data, seq_length): xs = [] ys = [] for i in range(len(data)-seq_length-1): x = data[i:(i+seq_length)] y = data[i+seq_length] xs.append(x) ys.append(y) return np.array(xs), np.array(ys) seq_length = 10 # 注意这里仅使用正常数据进行训练 X, y = create_sequences(normal_data, seq_length) # 将数据转换为PyTorch张量 X_torch = torch.Tensor(X) y_torch = torch.Tensor(y) # 定义LSTM模型 class LSTMModel(nn.Module): def __init__(self, input_size=1, hidden_layer_size=100, output_size=1): super().__init__() self.hidden_layer_size = hidden_layer_size self.lstm = nn.LSTM(input_size, hidden_layer_size) self.linear = nn.Linear(hidden_layer_size, output_size) def forward(self, input_seq): lstm_out, _ = self.lstm(input_seq.view(len(input_seq), 1, -1)) predictions = self.linear(lstm_out.view(len(input_seq), -1)) return predictions[-1] # 实例化模型、损失函数和优化器 model = LSTMModel() loss_function = nn.MSELoss() optimizer = torch.optim.Adam(model.parameters(), lr=0.001) # 训练模型 epochs = 50 for i in range(epochs): for seq, labels in zip(X_torch, y_torch): optimizer.zero_grad() y_pred = model(seq) single_loss = loss_function(y_pred, labels) single_loss.backward() optimizer.step() if i%10 == 0: print(f'当前轮数:{i},当前损失:{single_loss.item()}') # 使用模型进行预测 model.eval() test_predictions = [] with torch.no_grad(): for seq in X_torch: test_predictions.append(model(seq).item()) # 绘制结果,包括异常数据区间 plt.figure(figsize=(15, 6)) plt.plot(t, anomaly_data, label='Data with Anomalies') plt.plot(t[seq_length+1:], test_predictions, label='Predictions', alpha=0.7) plt.fill_between(t, -2, 4, where=(t >= 300) & (t <= 400), color='r', alpha=0.3, label='Anomaly Region') plt.legend() plt.show()


三、异常值处理
在常规的建模中,识别出异常数据之后通常有一下几种方法来处理异常值:
- 删除异常值:如果异常值数量不多,且对分析没有重要意义,可以直接将这些异常值从数据集中删除。这是最直接的处理方式,但可能会导致信息的丢失。
- 替换异常值:
- 用平均值、中位数或众数替换:对于连续变量,可以用非异常值的平均值或中位数替换异常值;对于分类变量,可以用众数替换。
- 用预测模型替换:构建一个预测模型,用其他变量的值预测异常值所在变量的值。
- 转换数据:
- 对数转换:对具有长尾分布的数据进行对数转换,可以缩小异常值与其他值之间的差距。
- Box-Cox转换:一种更通用的转换方法,可以减少数据的偏斜和异常值的影响。
- 分箱:将连续数据分成几个区间(箱),然后用箱的编号或箱中心值代替原始值。这种方法可以减少异常值的影响。
- 截断:将超过或低于某个阈值的异常值设置为阈值。这种方法可以保留异常值的存在,但减少其影响。
- 标记异常值:在模型中增加一个新的变量来标记数据点是否为异常值。这样做可以让模型自己决定如何处理异常值。
- 保留异常值:在某些情况下,异常值本身可能就是我们分析的重点,或者异常值对模型的影响不大。在这种情况下,可以选择保留异常值不做处理。
在异常检测建模中就自不必说了,异常值本就是我们的目的。